Программные инструменты и биологические базы данных для построения гипотез о взаимодействиях белков по результатам протеомного профилирования
Аннотация
Дата поступления статьи: 25.12.2013Авторами предложены два подхода к построению гипотез о взаимодействиях белков на основании результатов слежения за изменениями их уровня в ходе изучаемого биологического процесса. Первый подход основан на использовании машинного интеллектуального анализа биологических текстов, находящихся в свободном доступе в сети Интернет. Второй, связан с выполнением запросов к базам данных бинарных белок-белковых взаимодействий. Для уточнение контекста экспериментального воздействия в получаемых белковых сетях используется фильтрация сущностей этих сетей на основании необходимых онтологий базы Gene Ontology.
Ключевые слова: машинный интеллектуальный анализ текстов, база данных, белок-белковые взаимодействия, белковые сети
05.11.17 - Приборы, системы и изделия медицинского назначения
Современные методики протеомных экспериментов позволяют одновременно отслеживать изменения уровней сотен и тысяч белков [1-3]. В результате таких экспериментов удается установить основных молекулярных «действующих лиц» изучаемого процесса, однако не удается определить возможные межмолекулярные взаимодействия, лежащие в его основе. Целью настоящей работы была разработка подхода к построению молекулярных моделей реакций клеток на внешние воздействия, который, с одной стороны, базировался бы на данных, получаемых в ходе протеомного исследования, а с другой стороны, использовал бы потенциал постоянно пополняемых публичных баз данных о бинарных белок-белковых взаимодействиях. В совокупности такой подход позволил бы строить обоснованные гипотезы об объединении установленных «молекулярных участников» изучаемых процессов в сети молекулярных взаимодействий.
В качестве исходных данных, использовались результаты эксперимента по изучению реакции протеома нервной ткани речного рака на окислительный стресс, вызванный фотодинамическим воздействием [4]. Исходным набором данных служил список белков, демонстрировавших в ответ на фотодинамически генерируемый окислительный стресс, изменения уровня более чем на 25%. Для объединения белков исходного списка в гипотетическую сеть использовались два подхода к определению возможных бинарных белок-белковых взаимодействий: 1) программный интеллектуальный анализ аннотаций научных статей по молекулярной биологии, находящихся в базе PubMed (мы использовали демо-версию пакета Pathway Studio [5] от компании Ariadne Genomics); 2) запрос к специализированным базам данных белк-белковых взаимодействий (в большинстве случаев мы использовали базу BioGrid [6]) с помощью свободно распространяемого пакета Cytoscape [7].
Размер получаемых сетей молекулярных взаимодействий мог быть различным – в Pathway Studio это определялось количеством анализируемых статей, в Cytoscape можно было указать длину цепочки связей, обозначающих взаимодействия, по отношению к белкам входного списка (в среднем, получаемые сети состояли из 2-3 сотен белков, Рис.1).
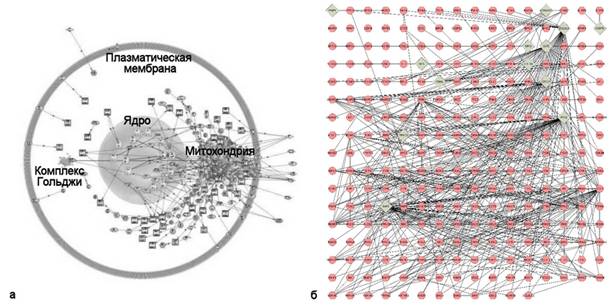
Рис. 1. – Сети белок-белковых взаимодействий, полученные с помощью программных средств Pathway Studio (a) и Cytoscape (b, серый цвет – белки исходного набора, розовый – их прямые партнеры по взаимодействиям).
В Pathway Studio нам не удалось обнаружить адекватных средств учета контекста экспериментального воздействия, вследствие чего в результирующую сеть попадали все бинарные взаимодействия между белками входного набора данных (и их соседями) вне зависимости от экспериментальных условий, в которых эти взаимодействия были обнаружены. Контекст нашего экспериментального воздействия - фотоокислительный стресс, был близок к одной из категорий (онтологий) биологических процессов базы GeneOntology (GO) [8], а именно - «реакция на окислительный стресс». Пакет Cytoscape, расширенный плагином Mosaic, позволил аннотировать сущности молекулярной сети, используя запрос онтологий к базе GO (Рис. 2а). Последующая фильтрация исходной сети с условием: «все белки входного набора + аннотированные, как участвующие в реакции на окислительный стресс» дала возможность получить искомую сеть (Рис. 2б, 3).

Рис. 2. – Cytoscape: (а) результат аннотирования белков полученной сети в соответствии с онтологиями базы GO (отдельные цвета соответствуют различным биологическим процессам), и последующей классификации (б) сущностей сети в соответствии с условием: «все белки входного набора (зеленый цвет) + аннотированные, как участвующие в реакции на окислительный стресс (желтый цвет)», прочие белки – красный цвет.
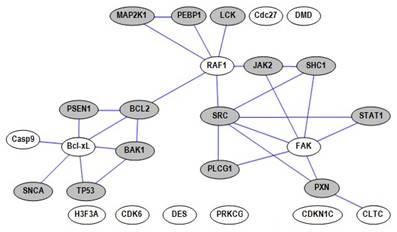
Рис. 3. - Прогнозируемая сеть белок-белковых взаимодействий, разворачивающихся в нервной ткани при фотоокислительном стрессе.
В сеть вошли белки входного набора данных (светлые фигуры), а также белки, которые одновременно участвуют в реакции клетки на окислительный стресс (по данным базы GO), и способны напрямую взаимодействовать с белками входного вектора (по данным базы BioGrid) – темные фигуры. Сокращенные названия белков даны в соответствии с нотацией, используемой в проекте proteinatlas.org
Для использования прогностических возможностей полученной модели можно обратить внимание на хабы, образованные классическими сигнальными белками Bcl-xL, RAF1, FAK, которые изменяли свои уровни в эксперименте. Любопытно, что они не входят в онтологию «реакция на окислительный стресс» базы GO. Учитывая также то, что и в литературе отсутствуют данные об участии этих белков в реакции клеток нормальной неопухолевой нервной ткани на фотодинамически-индуцируемый окислительный стресс, экспериментальная проверка роли этих белков может служить отправной точкой в дальнейшем изучении механизмов повреждения нервной ткани при окислительном стрессе. Аналогичным образом, используя данные протеомного профилирования, предложенный нами подход может быть использован для формулирования обоснованных гипотез о межмолекулярных взаимодействиях, разворачивающихся в ходе других биологических процессов или заболеваний.
Очевидно, что интерес к созданию автоматизированных средств поиска, анализа и обработки биомедицинской информации [9] будет неуклонно расти, а эффективность таких систем будет напрямую связана с появлением новых, более совершенных алгоритмов анализа текстов [10].
Литература:
1. Aebersold R, Mann M. Mass spectrometry-based proteomics [Текст] // Nature, 2003. - 422(6928). –P.198-207.
2. Sutandy FX, Qian J, Chen CS, Zhu H. Overview of protein microarrays [Текст] // Curr Protoc Protein Sci., 2013. – 27(27.1).
3. Yu X, Schneiderhan-Marra N, Joos TO. Protein microarrays for personalized medicine [Текст] // Clin Chem., 2010. - 56(3). – P.376-87.
4. Колосов М.С., Бибов М.Ю., Демьяненко С.В., Шарифулина С.А., Узденский А.Б. Протеомный анализ реакции нервной ткани рака на фотодинамически-индуцированный окислительный стресс [Текст] // Материалы XVI Международной конференции по нейрокибернетике. Ростов-на-Дону, 24-28 сентября, 2012, - C.20-23.
5. Nikitin A, Egorov S, Daraselia N, Mazo I. Pathway studio--the analysis and navigation of molecular networks [Текст] // Bioinformatics, 2003. - 19(16). – P.2155-2157.
6. Stark C, Breitkreutz BJ, Reguly T, Boucher L, Breitkreutz A, Tyers M. BioGRID: a general repository for interaction datasets [Текст] // Nucleic Acids Res., 2006. -34. –P.535-539.
7. Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks [Текст] // Genome Res., 2003. -13(11). –P.2498-2504.
8. Lomax J. Get ready to GO! A biologist's guide to the Gene Ontology [Текст] // Brief Bioinform., 2005. - 6(3). –P.298-304.
9. Дунин В.О., Егоров В.А. Проблемы создания интеллектуальных средств поиска, анализа и обработки биомедицинской информации [Электронный ресурс] // «Инженерный вестник Дона», 2012, №4 (ч.1). – Режим доступа: http://www.ivdon.ru/magazine/archive/n4p1y2012/1220 (доступ свободный) – Загл. с экрана. – Яз. Рус.
10. Красников И.А., Никуличев Н.Н. Гибридный алгоритм классификации текстовых документов на основе анализа внутренней связности текста [Электронный ресурс] // «Инженерный вестник Дона», 2013, №3. – Режим доступа: http://www.ivdon.ru/magazine/archive/n3y2013/1773 (доступ свободный) – Загл. с экрана. – Яз. Рус.